파일 형식을 알아보자! (FASTA, FASTQ, BAM, SAM)
생명정보학 분석에서 자주 만나는 파일 형식을 알아봅시다
안녕하세요, 한헌종입니다.
오늘은 생명정보학 분야에서 자주 만나게 되는 파일 형식이 어떤 게 있는지 알아봅시다.
생명정보학을 공부하면서 가장 많이 만나게 되는 파일 형식은 사실 tsv, csv일 것입니다.
이 형식들은 여러분이 잘 알고 계시는 ‘표’ 형태의 정보를 텍스트 파일로 나타낸 것입니다.
tsv 는 tab-separated values, 즉 ‘탭’으로 데이터를 나누어 적어놓은 파일이구요,
csv 는 comma-separated values, 즉 ‘쉼표’로 데이터를 나누어 적어놓은 파일입니다.
어려울 것 없이, 그냥 표라고 생각하시면 됩니다. tsv,csv 파일을 엑셀로 열면 아주 이쁘게 표로 표현된답니다.
 |
|---|
| csv 형태의 데이터는 표와 같습니다 |
세상의 모든 데이터가 tsv/csv 처럼 단순한 형태로 있으면 참 편할텐데, 그렇지가 않습니다.
생명정보학을 공부하시다 보면 아래에 소개드릴 특별한 파일 형식들을 많이 만나보시게 될 겁니다.
(사실 생각나는 대로 적어본 거라, 서로 연관성은 많이 없습니다.)
이 형식들에 익숙해지시면 데이터를 이해하고 분석하는 게 한결 수월해지실 겁니다.
그럼 한 번 알아볼까요?
FASTA
FASTA 파일은 특정 분자의 서열을 나타내는 데에 사용됩니다.
주로 Genome 의 각 chromosome 마다의 서열을 저장하는 데 쓰이죠.
혹은 gene/transcript/protein 각각의 염기서열 및 아미노산 서열을 저장할 때 사용되는 형식이에요.
FASTA 파일은 두 형태가 반복되는 형식입니다.
첫 부분은 그 아래에 나올 서열의 이름/ID 입니다. 항상 > (꺽쇠) 글자로 시작하죠.
다음에는 그 이름에 해당하는 서열이 나오게 됩니다.
보통 그 서열은 굉장히 깁니다. 염색체는 말할 것도 없고, 특정 유전자나 단백질도 몇백-몇만 글자로 표현해야 하죠.
그래서 이를 한 줄로 나타내지 않고, 보통 60글자씩 나눠서 여러 줄에 걸쳐 표현합니다.
 |
|---|
| FASTA 파일의 예시. 이름-서열-이름-서열 이 반복되네요 |
* 주의: FASTA 파일은 보통 엄청나게 크기 때문에, 가정용 컴퓨터에서 열어보지 않는 걸 추천합니다.
FASTQ
fastq 파일은 NGS (Next generation sequencing data) 의 결과를 저장하는 데 주로 쓰입니다.
NGS 실험을 진행하면, 그 결과로 cDNA library 서열을 읽어서 데이터로 얻을 수 있습니다.
즉, 각 cDNA library 의 염기서열을 알 수 있는거죠.
이 서열 하나를 ‘read’ 라고 하는데요.
fastq 파일은 여러 read 의 정보를 한 파일에 저장하게 됩니다.
보통 이 파일을 직접 사용하기보다, reference genome 에 align 한 뒤에 활용됩니다.
fastq 파일은 대부분의 연구에서 raw data, 즉 가공되지 않은 원본 파일로 여겨집니다.
그러니 분석하실 땐 fastq를 잘 백업해두셔서 잃어버리지 않도록 하세요!
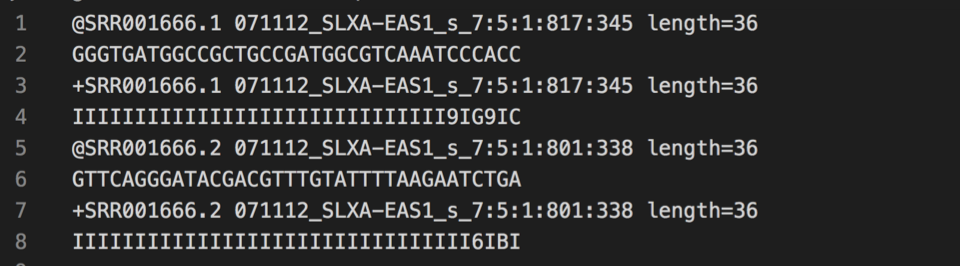
fastq 파일은 네 줄이 한 단위입니다.
위에서 말씀드린 것처럼 하나의 cDNA library 정보가 네 줄에 나눠서 표현되어 있는 거죠.
이들은 각각 다음 정보를 가지고 있습니다:
- sequence ID: @ 로 시작하며, 해당 서열의 이름을 나타냅니다.
- sequence: 실제로 읽은 염기서열 정보입니다.
- description: ‘+’ 글자로 시작하는데, + 하나만 있기도 하고 sequence ID를 넣거나 설명을 넣는 부분입니다.
- quality: 각 염기서열이 얼마나 정확히 읽혔는지를 나타냅니다. Phred quality score 라는 표현법을 사용합니다.
 |
|---|
| fastq 파일의 예시. 이름-서열-설명-품질 이 반복됩니다 |
* 주의: fastq 파일도 역시, 보통은 엄청나게 크기 때문에 가정용 컴퓨터에서 열어보지 않는 걸 추천합니다.
BAM, SAM
BAM 파일을 먼저 살펴볼까요.
BAM 은 binary alignment map 이라는 형식이에요.
이 파일은 위에서 설명한 fastq 파일을 reference genome에 align 했을 때 만들어지는 파일이죠.
즉, 각 cDNA library 조각이 reference genome 의 어느부분에서 나왔구나~ 하는 정보를 담았다는 거죠.
fastq 에서는 각 read 의 염기서열과 그 품질을 알 수 있다면, BAM 파일은 염기서열과 reference 에서의 위치정보 를 알 수 있습니다.
그렇지만 BAM 파일은 사람이 읽을 수 없는 파일입니다…
정보를 압축하기 위해 binary 형태로 저장된 파일이기 때문이죠.
다행히도 이 BAM 파일을 ‘볼 수 있게’ 해놓은 파일이 있습니다. 바로 SAM 파일입니다.
BAM 파일과 SAM 파일은 동일한 정보를 가지고 있고, 서로 변환이 가능합니다. (변환 방법은 다음에 설명드릴게요)
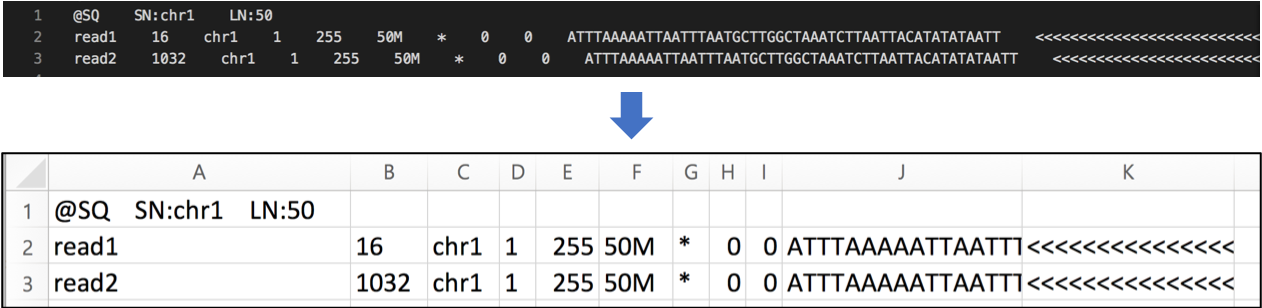
SAM 파일은 header 부분과 alignment 부분으로 이루어져 있습니다.
- header
header 부분은 파일에 대한 설명을 주는 부분입니다. @ 로 시작하는 라인들입니다. - alignment
alignment 부분이 각 read에 대한 alignment 정보를 제공하는 부분입니다.
필수적인 11개의 컬럼으로 이루어져 있고, 추가로 몇 개의 컬럼이 더 있을 수도 있습니다.
 |
|---|
| SAM 파일의 예시. 11개 컬럼이 너무 길어서 엑셀로 표시해보았어요 |
각 column의 설명은 다음과 같습니다:
- QNAME: read 이름
- FLAG: 2진수로 된 read alignment 에 대한 설명
- RNAME: refernce sequence 의 이름
- POS: reference sequence 에서 align 된 위치
- MAPQ: mapping quality. 즉 얼마나 정확히 align 되었는지.
- CIGAR string: alignment 정보를 표현한 문자열. Match, Gap 등의 설명을 각 염기마다 표현합니다.
- RNEXT: 다음 read 의 reference sequence 이름. 주로 paired end read 에 대한 분석을 위해 사용됩니다.
- PNEXT: 다음 read 의 align 된 위치. 주로 paired end read 에 대한 분석을 위해 사용됩니다.
- TLEN: Template length. paired-end read 둘의 left-end 부터 right-end 까지의 길이입니다.
- SEQ: segment sequence. 염기 서열을 나타냅니다.
- QUAL: Phread quality score 입니다.
와, 저도 이 11개 컬럼의 뜻을 모두 알지는 못했는데 이렇게 많은 정보가 있는 줄은 몰랐네요!
* 주의: SAM 파일도 역시, 읽을수는 있다고 하나 엄청나게 크기 때문에 가정용 컴퓨터에서 열어보지 않는 걸 추천합니다.
오늘은 FASTA, FASTQ, BAM/SAM 파일에 대해 알아봤습니다.
분석의 앞부분에서 꼭 마주치게 되는 형식들이죠.
평소에 잘 안다고 생각했던 것들인데, 자세히 들어가보니 내용이 무척 많네요.
언젠가 이 파일들이 분석 순서상으로 어떻게 연관되어 있는지도 설명드릴게요.
아, 그리고 이 외에도 널리 사용되는 파일 형식들이 많은데요, VCF/BED/GTF 등이 있어요.
이 형식들은 다음에 또 알아보도록 할게요.
그럼 다음시간에 만나요!