Human reference genome - GRCh37 과 GRCh38 에 대하여
안녕하세요 한헌종입니다.
오늘은 사람의 reference genome 에 대해 이야기하려 해요.
그리고 가장 널리 쓰이는 GRCh37 와 GRCh38 이 어떻게 다른지 이야기해보려 합니다.
이 내용은 다음 논문의 내용을 요약한 것입니다: Guo et al., Genomics, 2017
reference genome 을 간단히 설명하자면,
어떤 개체의 genome 서열을 대표하는 뉴클레오티드 서열 데이터를 말합니다.
이중 사람의 reference genome 도 존재하죠.
그런데, 사람마다 서열이 조금씩 다르기 때문에 사람의 reference genome 서열과 나의 서열은 약간 다를 것입니다.
사람의 reference genome 은 ‘대표’되는 서열을 만들기 위해 여러명이 기증한 샘플로 만들어낸 거죠.
reference genome 을 만드는 기관은 GRC (Genome Reference Consortium) 입니다.
그리고 여기서 만든 reference genome 중 사람 (h) 의 버전 38 에 해당하는 것이 바로 우리가 흔히 보는 GRCh38 인 겁니다.
그럼 reference genome 은 어떻게 만들까요?
genome 을 알아내기 위해서는 ‘genome assembly’ 라는 과정을 거쳐야 합니다.
이는 짧은 서열인 read 들을 서로 공통된 부분끼리 붙여서 점점 길게 붙여나가는 과정이죠.
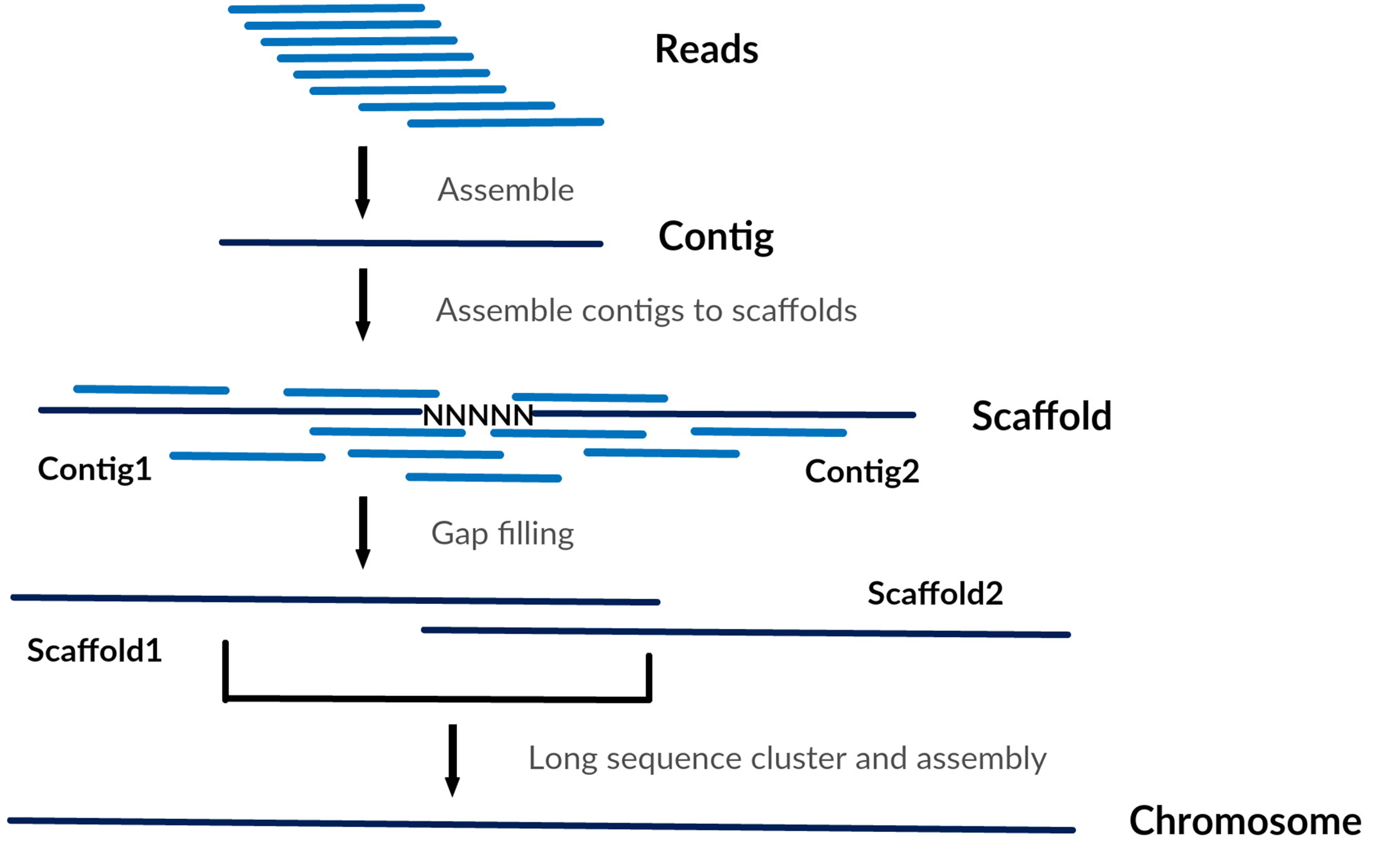
아래 그림에 간단히 설명되어 있습니다.
 |
|---|
| 출처: Guo et al., Genomics (2017) |
그림을 보시면 Reads 를 붙여서 contig 를 만들고, 다시 contig 들을 붙여서 scaffold 를 만들고,
마지막으로 이 scaffold 들을 붙여 염색체 하나를 이루게 되는 것입니다.
현재 기술로는 ‘완벽하게 정확한’ genome sequence 를 알아내는 것이 힘듭니다.
그래서 계속 수정에 수정을 거쳐 더 정확한 reference genome 버전이 만들어지는 것이죠.
완벽한 genome sequence 를 만들기 힘든 이유를 간단히 말하자면,
Error-prone short reads 를 통해 error-prone repeat 을 가진 large sequence 를 조립하는 것이 매우 힘들기 때문입니다.
그래서 가장 최신 버전인 GRCh38 조차도 아직 서열을 알 수 없는 Gap 부분이 많이 있습니다.
최근 GRC 에서 이런 에러와 gap 을 해결한 human reference genome 을 만들었다고 하네요.
얼마전까지만 해도 완벽하게 만들 수 없다고 생각했는데 말이죠.
관심 있으신 분들은 이 논문을 참고해보세요: 링크
GRCh37 과 GRCh38 의 차이점
이제 본론으로 돌아와서, GRCh37 과 GRCh38 이 어떻게 다른지 이야기 해볼게요.
2009 년, GRC 는 HG19 라고도 불리는 새로운 버전의 human reference genome GRCh37 을 발표합니다.
이 때는 일루미나의 high-throughput sequencing 기술을 사용했습니다.
몇년 뒤인 2013년, GRC 는 또 다른 reference genome 인 GRCh38 을 발표하게 됩니다.
이 때는 더 많은 donor sample 을 사용했으며, Sanger sequencing 기술을 사용해 더 길고 정확한 reads 를 만들었습니다.
당연히 더 정확한 서열을 만들어냈겠죠?
실제로 GRCh37 이 가지고 있던 문제들을 1000개 이상 해결했다고 합니다.
그럼 이 두 가지 reference genome 이 실제 분석상으로 어떤 차이가 있을까요?
논문에서 언급한 두 reference genome 의 차이점을 아래 표에 간단히 정리해보았어요.
| Feature | GRCh37 | GRCh38 | change |
|---|---|---|---|
| Total nucleotides | 3,095,677,412 | 3,088,269,832 | 7,407,580 ▼ |
| # of “N” (gap / unannotated regions) | 234,350,281 | 150,630,719 | 83,719,562 ▼ |
| GC contents | 1,170,371,008 | 1,200,551,672 | 30,180,664 ▲ |
| Exome size | 75,231,228 | 95,505,476 | 20,274,248 ▲ |
| # of total exon | 327,058 | 457,748 | 130,690▲ |
| median # of exons per gene | 13 | 19 | 6▲ |
| median # of nucleotides per exon | 140 | 146 | 6▲ |
결과를 보시면, 먼저 전체 nucleotide 수는 0.2% 정도 줄어들었습니다.
그런데 이보다 더 중요한 건, 바로 “N” 으로 표시된 gap 이 줄어들었다는 겁니다.
gap 은 centromere 나 telomere 같이 알아내기 힘든 영역을 일단 N 이라고 표시한 부분인데요,
GRCh37 보다 GRCh38 에서는 이런 N 의 수가 8천만개 이상 줄었다는 것을 알 수 있습니다.
여기서 또 주목할 점은 바로 늘어난 Exome 의 크기입니다.
GRCh37 에 비해 GRCh38 에서 2천만 base pair 만큼, 약 26% 나 늘어난 것을 알 수 있습니다.
Exon 의 개수도 32만 개에서 45만 개로 40% 정도 늘어났습니다.
Exome 영역은 genome annotation 에 큰 영향을 주기 때문에, 이렇게 Exome 영역이 늘어났다는 것은 우리가 genome 에서 더 많은 정보를 얻을 수 있게 되었다는 뜻입니다.
실제 분석에 사용했을 때도 좋을까?
위의 정리된 표만 봐서는 아직 GRCh38 이 얼마나 더 좋은건지 알기 힘들 수 있습니다.
그래서 위 논문에서는 실제 Alignment, Variant call 등의 분석을 진행했을 때 얼마나 좋아지는지도 연구해놓았어요.
1. Alignment rate 비교
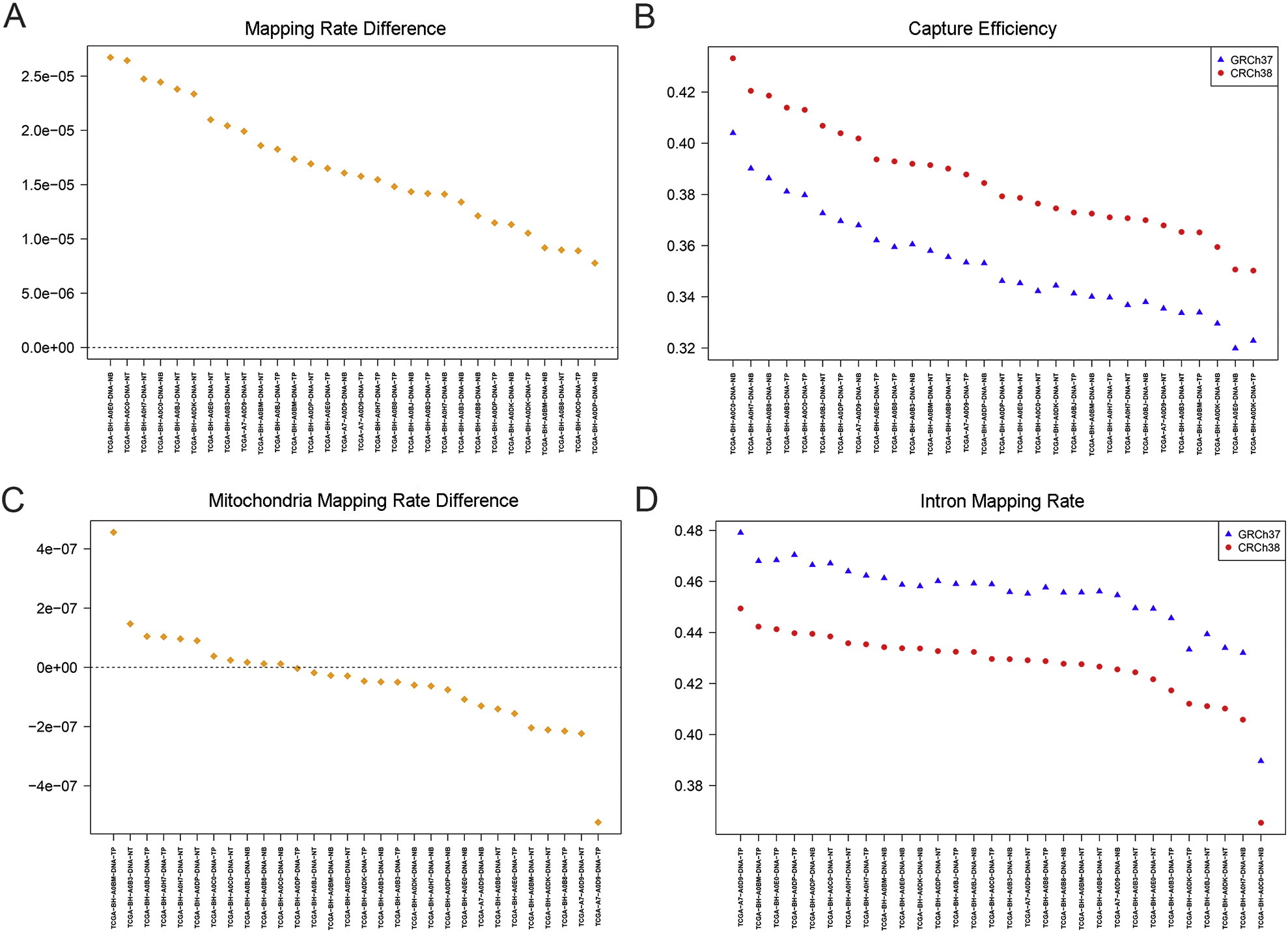
먼저 해본 것은 30개 샘플에서 얻은 WES 데이터를 각 reference genome 에 alignment 해본 거에요.
아래 그림에는 이 샘플들의 Mapping rate, Capture efficiency 등을 비교한 결과가 정리되어 있습니다.
 |
|---|
| 출처: Guo et al., Genomics (2017) |
이 결과를 보시면, 먼저 mapping 자체는 두 reference genome 에서 큰 차이가 없는 걸 알 수 있어요.
대신, Capture efficiency, 즉 Exome 에 mapping 된 read 비율이 3.2 % 정도 증가했습니다 (그림 B).
또한, Intron mapping rate 의 비율은 2.7 % 정도 줄어들었다고 하는군요 (그림 D).
이는 GRCh38 에서 exome 이 더 잘 정의되어 있기 때문에 생긴 결과라고 볼 수 있습니다.
mapping rate 가 높아지면 더 정확한 분석을 할 수 있으니, GRCh38 을 사용하는게 더 좋을 것입니다.
2. Variant calling 비교
alignment 를 진행한 뒤, 논문에서는 30 샘플의 WES 데이터에 어떤 변이가 각각 얼마나 있는지를 알아봤다고 합니다.
alignment 결과를 보고 reference genome 과 비교해서 변이를 찾는 과정을 variant calling 이라고 합니다.
과연 GRCh37 을 썼을 때와 GRCh38 을 썼을 때 결과가 어떻게 달라졌을까요?
아래 표는 30개 WES 샘플에서 발견한 SNV 변이의 종류와 그 수를 나타낸 거에요.
| Categories | Grch37 | Grch38 | Difference |
|---|---|---|---|
| Downstream | 29,647 | 29,841 | 194 |
| Exonic | 46,524 | 46,363 | − 161 |
| Exonic; splicing | 11 | 11 | 0 |
| Intergenic | 2,627,378 | 2,585,510 | − 41,868 |
| Intronic | 1,651,974 | 1,642,466 | − 9508 |
| Ncrna_exonic | 14,881 | 15,107 | 226 |
| Ncrna_exonic; splicing | 5 | 5 | 0 |
| Ncrna_intronic | 215,486 | 228,021 | 12,535 |
| Ncrna_splicing | 77 | 76 | − 1 |
| Splicing | 146 | 138 | − 8 |
| Upstream | 26,203 | 25,884 | − 319 |
| Upstream; downstream | 971 | 1213 | 242 |
| Utr3 | 36,065 | 36,031 | − 34 |
| Utr5 | 7076 | 7176 | 100 |
| Utr5; Utr3 | 17 | 17 | 0 |
| Total | 4,656,461 | 4,617,859 | − 38,602 |
가장 마지막에 있는 Total 을 보시면, 발견한 SNV 변이의 수가 GRCh38 에서 38,602 개 만큼 줄었어요.
그리고 Exonic, 즉 exon 영역에 발생한 변이의 수 역시 161 개 줄었네요.
이렇게만 보면 “GRCh38 을 써서 더 적은 변이를 발견했으니 안좋은 거 아닐까?” 라고 생각할 수 있어요.
하지만 다음 표를 보시면, GRCh38 에서 더 많은 정보를 얻을 수 있었다는 걸 알 수 있습니다.
| Categories | GRCh37 | GRCh38 | Difference |
|---|---|---|---|
| Nonsynonymous | 22,372 | 22,538 | 166 |
| Stopgain | 223 | 231 | 8 |
| Stoploss | 27 | 27 | 0 |
| Synonymous | 23,178 | 23,270 | 92 |
| Unknown | 735 | 308 | − 427 |
| total | 46,535 | 46,374 | − 161 |
이 표는 exon 영역 및 splicing 영역에서 일어난 SNV 들의 종류를 보여주고 있습니다.
GRCh38 을 사용했을 때 전체 SNV 개수는 줄었지만, nonsynonymous 변이는 늘고 unknown 변이는 줄었어요.
즉, 아미노산 변화를 일으키는 변이를 더 찾을 수 있었고, 뭔지 알 수 없는 변이의 수가 줄었다는 거죠.
이렇게 GRCh38 을 사용했을 때 더 많은 정보를 얻을 수 있게 되었다는 것을 논문을 통해 알 수 있었습니다.
결론: GRCh38 을 사용하는 것이 더 좋다
논문의 결론 부분에서는 다음과 같이 언급하고 있습니다.
“… we can safely conclude that GRCh38 is an improvement over GRCh37 and these improvements resulted in more accurate genomic analysis results. …”
유전체 분석을 더 정확히 할 수 있다는 관점에서 GRCh38 이 GRCh37 보다 더 향상되었다는 것입니다.
물론 아직 많은 연구기관에서 GRCh37 데이터를 생산 및 분석하고 있지만, 결국에는 GRCh38 혹은 그보다 더 발전된 버전의 reference genome 을 사용해야 하고, 머지않아 그렇게 될 겁니다.
이렇게 human reference genome 두 종류와, 그들이 어떻게 다른지, 그리고 분석에 어떤 영향을 미칠 수 있는지를 간단히 알아보았어요.
더 자세한 내용은 논문에 있으니 한번 참고해보세요! 링크: Guo et al., Genomics, 2017
그럼 다음 시간에 만나요!