BAM/SAM 의 flag 정보, samtools flagstat 설명
안녕하세요 한헌종입니다.
오늘은 bam / sam 파일에 있는 flag 에 대해, 그리고 이 flag 정보를 요약해 보여주는 samtools flagstat 결과에 대해 알아보겠습니다.
BAM / SAM 파일의 flag 란?
flag 란 무엇일까요?
flag 는 bam / sam 파일에서 각 read alignment 에 대한 설명을 숫자로 나타낸 것이에요.
이 flag 는 0 ~ 4095 사이의 숫자인데요, SAM 파일의 두 번째 컬럼이 가지고 있는 숫자를 말해요.
이전 포스트에 있는 그림을 다시 보여드릴게요.
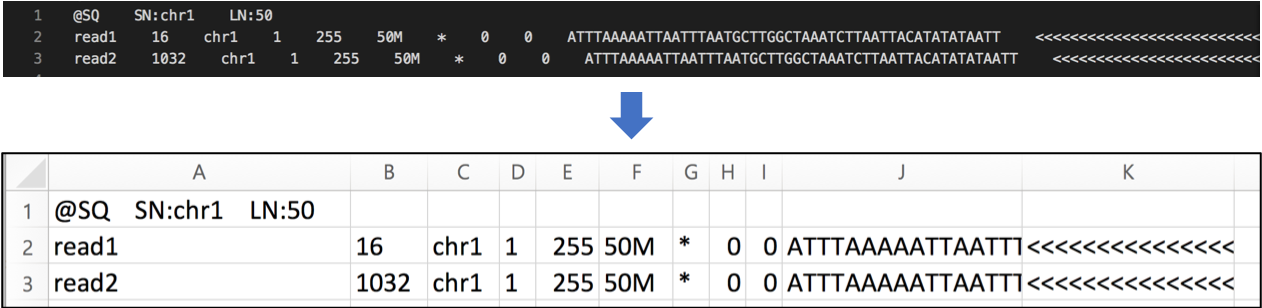 |
|---|
| SAM 파일의 예시. 2 번째 컬럼 (B) 에 적힌 숫자가 각 read 에 대한 flag 입니다. |
read1 의 flag 는 16, read2 의 flag 는 1032 이군요.
그럼 이 숫자가 무엇을 뜻하는 걸까요?
이걸 알아내려면 flag 를 이진수로 나타내봐야 합니다.
flag 를 이진수로 나타내기
flag 는 0~4095 사이의 숫자라고 했죠?
이를 2진수로 바꾸면 최대 12자리 이진수가 됩니다. (이때 빈 자릿수는 0으로 채워서 12자리를 맞춰줍니다.)
예를 들어 16 이라는 flag 값을 2진수로 나타내면 000000010000 가 되구요,
1032 라는 flag 값을 2진수로 나타내면 010000001100 가 됩니다.
왜 flag 값을 12자리의 2진수로 바꾸는 걸까요?
이는 read alignment 가 가질 수 있는 12가지 특성 각각이 있느냐, 없느냐를 표현하기 위해서에요.
2진수로 바꾼 flag 값에서 각 자리수는 각 특성을 나타내고, 1 이 의미하는 것은 해당 특성을 가지고 있다는 뜻이에요.
예를 들어 1032 라는 flag 값을 이진수로 나타낸 010000001100 를 보시죠.
이 숫자를 보면, 뒤에서 3번째 / 4번째 / 11번째 숫자가 1이고, 나머지는 모두 0이네요. (뒤에서부터 세어야 합니다)
즉, 이 read alignment 는 12가지 특성 중 3번째 / 4번째 / 11번째 특성을 가지고 있다는 뜻이에요.
flag 의 각 자리수는 어떤 특성을 가리키나요?
다음 표에는 각 자리수가 의미하는 바를 정리해보았어요.
이는 SAM format specification 문서에 있는 표를 참고했습니다. (SAM format specification 문서 링크)
| bit | 2진수 | 16진수 | 의미 |
|---|---|---|---|
| 1 | 000000000001 | 0x1 | template having multiple segments in sequencing |
| 2 | 000000000010 | 0x2 | each segment properly aligned according to the aligner |
| 4 | 000000000100 | 0x4 | segment unmapped |
| 8 | 000000001000 | 0x8 | next segment in the template unmapped |
| 16 | 000000010000 | 0x10 | SEQ being reverse complemented |
| 32 | 000000100000 | 0x20 | SEQ of the next segment in the template being reverse complemented |
| 64 | 000001000000 | 0x40 | the first segment in the template |
| 128 | 000010000000 | 0x80 | the last segment in the template |
| 256 | 000100000000 | 0x100 | secondary alignment |
| 512 | 001000000000 | 0x200 | not passing filters, such as platform/vendor quality controls |
| 1024 | 010000000000 | 0x400 | PCR or optical duplicate |
| 2048 | 100000000000 | 0x800 | supplementary alignment |
그러면 1032 라는 flag 는 무엇을 뜻할까요?
아까 말씀드렸듯, 이 read alignment 는 12가지 특성 중 3번째 / 4번째 / 11번째 특성을 가지고 있다는 뜻이므로
- segment unmapped: 이 read 는 alignment 가 되지 않은 read 라는 뜻입니다.
- next segment in the template unmapped: 이 read 는 paired-end 인데, pair 를 이루는 다른 한쪽의 read 도 alignment 가 되지 않았다는 뜻입니다.
- PCR or optical duplicate: 이 read 는 PCR 과정에서 만들어진 중복 read 라는 뜻입니다.
따라서, flag 가 1032 라는 것은 이 read 가 1) unmapped 이고 2) pair 역시 unmapped 이며 3) duplicate read 라는것을 뜻해요.
이렇게 flag 로 read 의 특성을 알 수 있답니다.
samtools 의 flagstat 보는 방법
이전 포스트에서 설명드렸듯, samtools 의 여러 기능 중 하나는 flagstat 이라는 명령어로 특정 read 들의 요약된 정보를 보는거에요.
(이전 samtools 명령어에 대한 링크 참조)
다음 예시를 보세요.
$ samtools flagstat example.bam
4579959 + 0 in total (QC-passed reads + QC-failed reads)
0 + 0 secondary
0 + 0 supplementary
371078 + 0 duplicates
4532991 + 0 mapped (98.97% : N/A)
4569463 + 0 paired in sequencing
2284756 + 0 read1
2284707 + 0 read2
4413160 + 0 properly paired (96.58% : N/A)
4475527 + 0 with itself and mate mapped
46968 + 0 singletons (1.03% : N/A)
21515 + 0 with mate mapped to a different chr
15292 + 0 with mate mapped to a different chr (mapQ>=5)
예시로 만든 bam 파일의 flagstat 결과에요.
여기 나온 특성들은 위의 flag 에 설명드린 특성이랑 맞는것도 있는것 같고, 아닌것도 있는 것 같죠?
samtools 문서를 보면 아래처럼 나와있습니다. 링크
| 결과 이름 | 설명 |
|---|---|
| secondary | 0x100 bit set |
| supplementary | 0x800 bit set |
| duplicates | 0x400 bit set |
| mapped | 0x4 bit not set |
| paired in sequencing | 0x1 bit set |
| read1 | both 0x1 and 0x40 bits set |
| read2 | both 0x1 and 0x80 bits set |
| properly paired | both 0x1 and 0x2 bits set and 0x4 bit not set |
| with itself and mate mapped | 0x1 bit set and neither 0x4 nor 0x8 bits set |
| singletons | both 0x1 and 0x8 bits set and bit 0x4 not set |
| with mate mapped to a different chr | 0x1 bit set and neither 0x4 nor 0x8 bits set and MRNM not equal to RNAME |
| with mate mapped to a different chr (mapQ>=5) | 0x1 bit set and neither 0x4 nor 0x8 bits set and MRNM not equal to RNAME and MAPQ >= 5 |
왜인지 몰라도 16진수로 써놨어요.
밑에는 16진수 표기가 아닌 bit 와 2진수에 따라서 좀 더 보기 편하게 표를 정리해봤어요.
| 결과 이름 | bit | 2진수 | 설명 |
|---|---|---|---|
| secondary | 256 | 000100000000 | secondary alignment |
| supplementary | 2048 | 100000000000 | supplementary alignment |
| duplicates | 1024 | 010000000000 | PCR or optical duplicate |
| mapped | 4 | 000000000100 | NOT segment unmapped |
| paired in sequencing | 1 | 000000000001 | template having multiple segments in sequencing |
| read1 | 1 AND 64 | 000000000001 000001000000 |
- template having multiple segments in sequencing - the first segment in the template |
| read2 | 1 AND 128 | 000000000001 000010000000 |
- template having multiple segments in sequencing - the last segment in the template |
| properly paired | 1 AND 2 AND NOT 4 | 000000000001 000000000010 000000000100 |
- template having multiple segments in sequencing - each segment properly aligned according to the aligner - NOT segment unmapped |
| with itself and mate mapped | 1 AND NOT 4 AND NOT 8 | 000000000001 000000000100 000000001000 |
- template having multiple segments in sequencing - NOT segment unmapped - NOT next segment in the template unmapped |
| singletons | 1 AND 8 AND NOT 4 | 000000000001 000000001000 000000000100 |
- template having multiple segments in sequencing - next segment in the template unmapped - NOT segment unmapped |
| with mate mapped to a different chr | 1 AND NOT 4 AND NOT 8 AND MRNM not equal to RNAME |
000000000001 000000000100 000000001000 |
- template having multiple segments in sequencing - NOT segment unmapped - NOT next segment in the template unmapped - mate reference name != references sequence name |
| with mate mapped to a different chr (mapQ>=5) | 1 AND NOT 4 AND NOT 8 AND MRNM not equal to RNAME AND MAPQ >= 5 |
000000000001 000000000100 000000001000 |
- template having multiple segments in sequencing - NOT segment unmapped - NOT next segment in the template unmapped - mate reference name != references sequence name - mapping quality >= 5 |
저도 각 결과값이 뭔지 세세히는 알지 못했는데, 이렇게 정리하고 나니 어떤 flag 를 사용해 만들어진 결과인지 알 수 있었습니다.
오늘은 bam/sam 에 있는 flag 에 대한 설명과, flagstat 결과가 ‘실제로’ 무엇을 의미하는지 살펴봤습니다.
표로 정리해봤는데 더 복잡해 보이기도 하네요 ^^;
그러나 그냥 결과를 보고 ‘그런가보다’ 하는것보다, 실제로 어떤 걸 가리키는 값인지 이해하면 더 좋겠죠?
그럼 다음에 만나요!